t-sne(Sklearn)の使い方とプロット(Seaborn)
Web上で見る例がMNISTなどのデータセットで書かれている場合が多いので,もっと単純な場合を例にt-sneの使い方をメモしておく。 また,その描画で手こずったのでそのメモもしておく。描画はMatplotlibを使って各クラスを色分けする方法で手こずった。ただ散布図を描いて,各クラスのラベルで色やマーカの形を変えたかっただけ。Excelなら何も悩まない描画なのに,Matplotlibではやたらと手こずる。colorbar, cmapなど色々調整方法を調べたが,結局,Seabornを使うのが覚えやすい(忘れにくい,思い出しやすい)と自分の中では結論。Seabornは最初はPandasのDataframeに変換するのが面倒だ,とも思ったが,Excelとやっていることは一緒なので良しとする。
t-SNEの使い方 in Sklearn
覚えておくことは,fit_transformに渡すデータの形式。渡すデータは,データ数をN,(一般的に高次元な)次元をDとすると,(N,D)のshapeのndarray。これが,(N, 2 or 3)に変換される。
描画
Seabornはscatterplotで散布図が描ける。データはPandasのデータフレームとして渡して,x,yのカラム名を渡す。特に,クラスで色分けする場合にはhueにラベルを渡す。t-sneの結果であるndarrayをPandas.DataFrameに変換する方法は幾つかあると思うが,カラム名とそのカラムのデータをDictionaryとして渡す形式が一番覚えやすい。ほかは渡し方でIndex方向とColumn方向が意図と逆になるなどが煩わしい。
例
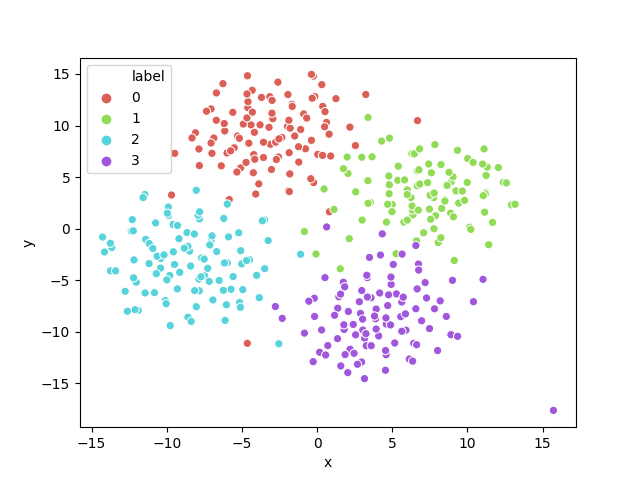
ここでの例は,4つのクラスタに別れるようなデータを作っている。64次元の空間に各クラスタのデータ(d1, d2, d3, d4)がそれぞれ中心位置をずらして正規乱数で生成されている。
import matplotlib.pyplot as plt import numpy as np import pandas as pd import seaborn as sns from sklearn.manifold import TSNE # Psedu dataset, (4 cluster). N = 100 # the number of data for each cluster D = 64 # space-dimension d1 = np.random.randn(N, D) + \ np.array([1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, # 16 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0]) l1 = np.array(['0']*N) d2 = np.random.randn(N, D) + \ np.array([0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0]) l2 = np.array(['1']*N) d3 = np.random.randn(N, D) + \ np.array([0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0]) l3 = np.array(['2']*N) d4 = np.random.randn(N, D) + \ np.array([0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1]) l4 = np.array(['3']*N) labels = np.concatenate([l1, l2, l3, l4], axis=0) dataset = np.concatenate([d1, d2, d3, d4], axis=0) # Reduce dimension by t-sne latent_vecs = TSNE(n_components=2).fit_transform(dataset) # Plot dataset with Seaborn df = pd.DataFrame(data={'x': latent_vecs[:, 0], 'y': latent_vecs[:, 1], 'label': labels}) sns.scatterplot(data=df, x='x', y='y', hue='label', palette=sns.color_palette('hls', 4)) plt.savefig('tsne.png') plt.show()